

| Kl. pneumoniae | Enterobacter spp | Ps. Aeruginosa | A. baumanii | S. aureus | |||||||||||
| No | %R | %I | No | %R | %I | No | %R | %I | No | %R | %I | No | %R | %I | |
| WARDS | 496 | 12,3 | 1,8 | 521 | 16,3 | 2,7 | 993 | 27,8 | 3,4 | 299 | 76,6 | 2,0 | 688 | 27,0 | 2,2 |
| I.C.U | 149 | 55,7 | 7,4 | 96 | 39,6 | 7,3 | 421 | 66,5 | 2,9 | 238 | 92,4 | 0,9 | 273 | 68,9 | 1,9 |
| OUTPATIENTS | 66 | 3,3 | 0,0 | 63 | 3,3 | 1,7 | 95 | 11,6 | 2,1 | 2 | 0 | 0 | 110 | 5,6 | 2,6 |
B. Percent resistance to ciprofloxacin by hospital (only wards are included).
Data from 9 hospitals are included.
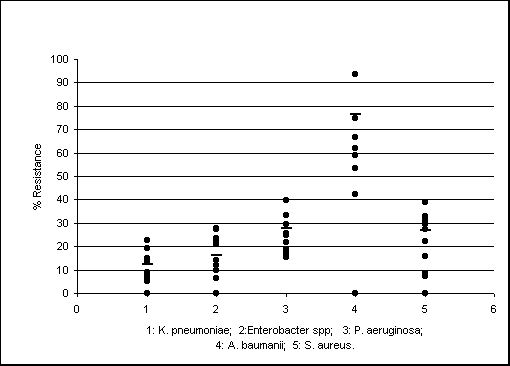
C. Resistance phenotypes of the ciprofloxacin resistant clinical isolates
| K. pneumoniae | Enterobacter spp | P. aeruginosa | A. baumanii | S. aureus | |||||
| No of isolates: 68 | No of isolates : 74 | No of isolates:217 | No of isolates:339 | No of isolates 413 | |||||
| Phenotype | % | Phenotype | % | Phenotype | % | Phenotype | % | Phenotype | % |
| ID1G2X* | 30,9 | D 2X | 36,5 | PD1G2N | 24 | DG2 | 26 | OEXG | 44,6 |
| ID1G2XN | 16,2 | DG2X | 27 | PD1G2 | 11,5 | SDG2 | 13,9 | OE G | 32,9 |
| ID1 2X | 16,2 | D X | 9,5 | P 1G2 | 11,5 | SDG | 12,1 | OE | 8,2 |
| D1G2X | 7,4 | DG X | 8,1 | P 1G2N | 10,6 | SD 2 | 9,7 | O XG | 5,3 |
| D1 2X | 7,4 | D1G2N | 7,4 | D 2 | 8,8 | ||||
| DG2N | 8 | ||||||||
| DG | 8 | ||||||||
| Other | 22,1 | Other | 18,9 | Other | 35 | Other | 13,6 | Other | 9 |
| ALL | 100 | ALL | 100 | ALL | 100 | ALL | 100 | ALL | 100 |